CUT&Tag-IT® Spike-In Control, R-loopの概要
Cleavage Under Targets and Tagmentation (CUT&Tag)は、ヒストン修飾の局在をゲノムワイドにプロファイリングするための強力な方法として登場しました。アクティブ・モティフは、この方法を応用してDNA-RNAハイブリッドからなるR-loopをゲノムワイドに検出するキットを開発しました。CUT&Tag-IT® R-loop Assay Kitは、当社のDNA-RNA hybrid (Clone S9.6)抗体を含んでおり、最適化されたプロトコルを使用してゲノムワイドなR-loop解析をすることができます。
次世代シーケンスでは、各サンプルの細胞数やリード数のばらつきにより、生物学的差異が不明瞭になる場合がありますが、アクティブ・モティフのCUT&Tag標準化戦略をCUT&Tag-IT R-loop Assayに適用することで、サンプル間の真の違いを明らかにすることができます。
CUT&Tag-IT® Spike-In Control, R-loopの特長
- CUT&Tag-IT R-loop Assay Kitを利用したサンプル間のデータ解析に最適。
- Spike-In Nucleiを解析したいサンプルに加え、DNA-RNA Hybrid抗体(Clone S9.6) とSpike-In Antibodyを使ってCUT&Tagを実行するだけ。
- 標準化ファクターを得ることにより、真のサンプル間の差異を見出すことが可能。
CUT&Tag-IT® Spike-In Control, R-loopの構成品
- CUT&Tag-IT Spike-In Antibody, Mouse, store at -20°C
- CUT&Tag-IT Spike-In Nuclei, R-loop, store at -80°C
CUT&Tag-IT® Spike-In Control, R-loopのデータ
アクティブ・モティフのCUT&Tag標準化戦略1は、哺乳類サンプルとの交差反応性がないSpike-In Antibodyを用いるため、あらゆる哺乳類CUT&Tagアッセイ反応に適用できます。CUT&Tag反応ごとに使用する凍結保存されたショウジョウバエ由来の核と抗体の量は、ショウジョウバエのリードが全シーケンスリードの 5~10%となるように最適化する必要がある場合があります。
R-loopを解析対象とする CUT&Tagでは、Spike-In : Test Sampleの比率を1 : 10とすることが推奨されます。このアプローチの有用性を実証するために、異なる細胞数でCUT&Tag反応を実施し、DNA-RNAハイブリッドのR-loopレベルの違いを調べました。各実験では、凍結保存した細胞数の異なるヒトK562 細胞(500,000、375,000、250,000、125,000、62,500および31,250個)と50,000個の凍結保存したショウジョウバエの核と組み合わせました (図 1)。
いずれの細胞数も、各サンプルはそれぞれduplicate用意し、CUT&Tag R-loop Spike-Inアッセイを行い、DNA-RNAハイブリッドを評価しました。ライブラリーは定量した後、サンプルあたり4,000~5,000 万リードでシーケンシングを実施しました。しかし、各サンプルのリード数が同等になるようにシーケンスすると、各サンプル量の初期値の違いが隠れてしまいます(図2)。したがって、細胞量の初期値の違いを明らかにするには、spike-inによる標準化が必要になります2。標準化では、ショウジョウバエのリード数が最も少ないサンプルの値を使用して各サンプルの標準化係数を算出し、それに応じて各サンプルにおけるヒトのリード数をダウンサンプリングしました。標準化されたヒトサンプルのリード数を取得した後、標準的な CUT&Tag 解析パイプラインを使用してbigWigのピークコールを行いました。標準化された結果は元の細胞数と相関があるため、実験サンプル間の差異を明らかにできます (図 3)。
References
- Egan, B. et al. (2010) PLoS ONE. 11(11): e0166438
- Taruttis et al. (2017) Biotechniques 62:53-61
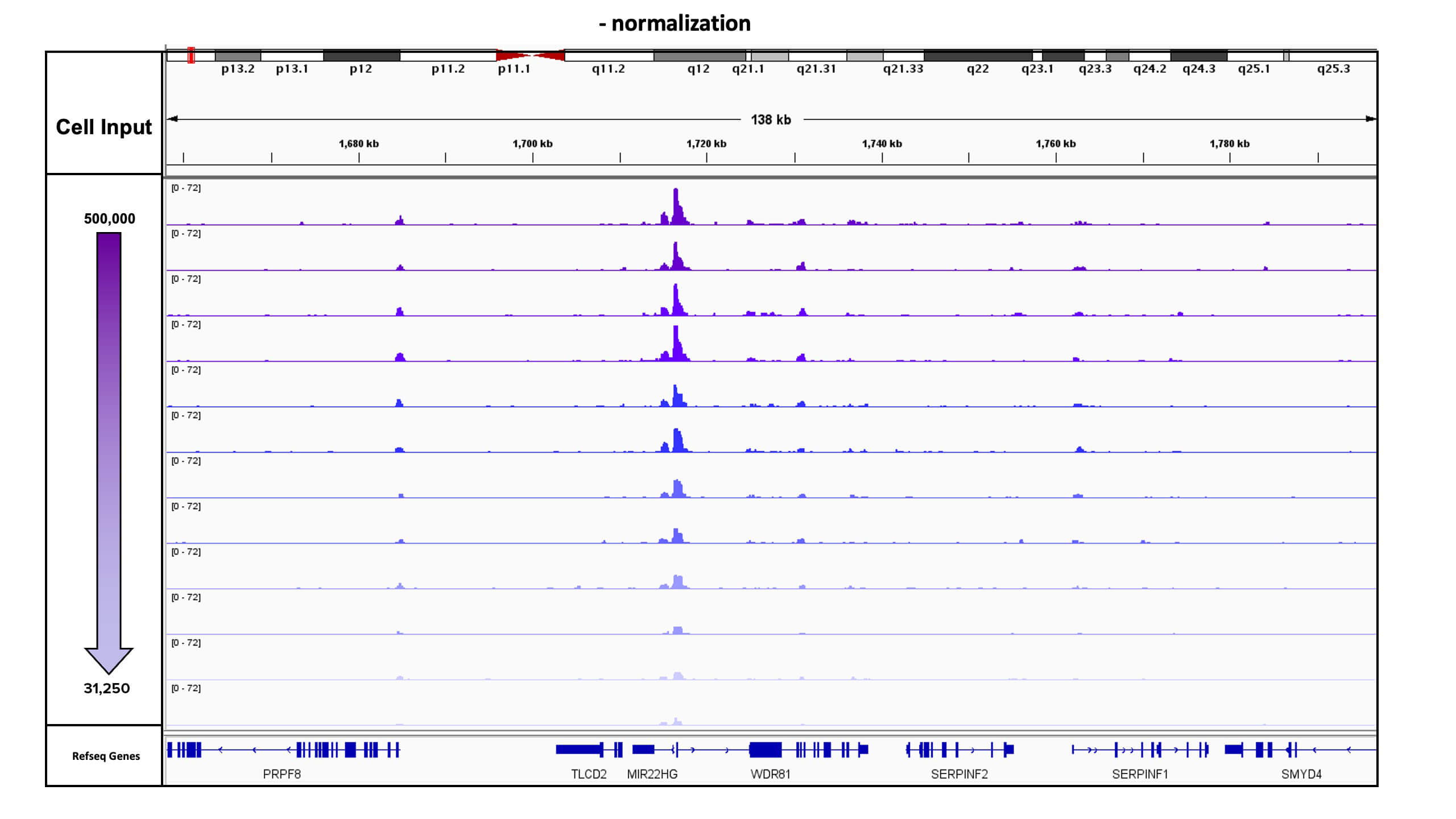
図1: 異なる数のK562細胞に対し、CUT&Tag-IT R-loop Assayを実施した例(Spike-In標準化なしの場合)
この実験では、モック処理のSpike-Inとして50,000個のdrosophila (ショウジョウバエ)の核を使用した。各サンプルには500,000個から31,250個の凍結保存したK562細胞を用いてduplicateで実施した。この図は、duplicateで実施した標準化なしのトラックをすべて示している。ブラウザの示す遺伝子座はchr17:1,657,948-1,796,865である。
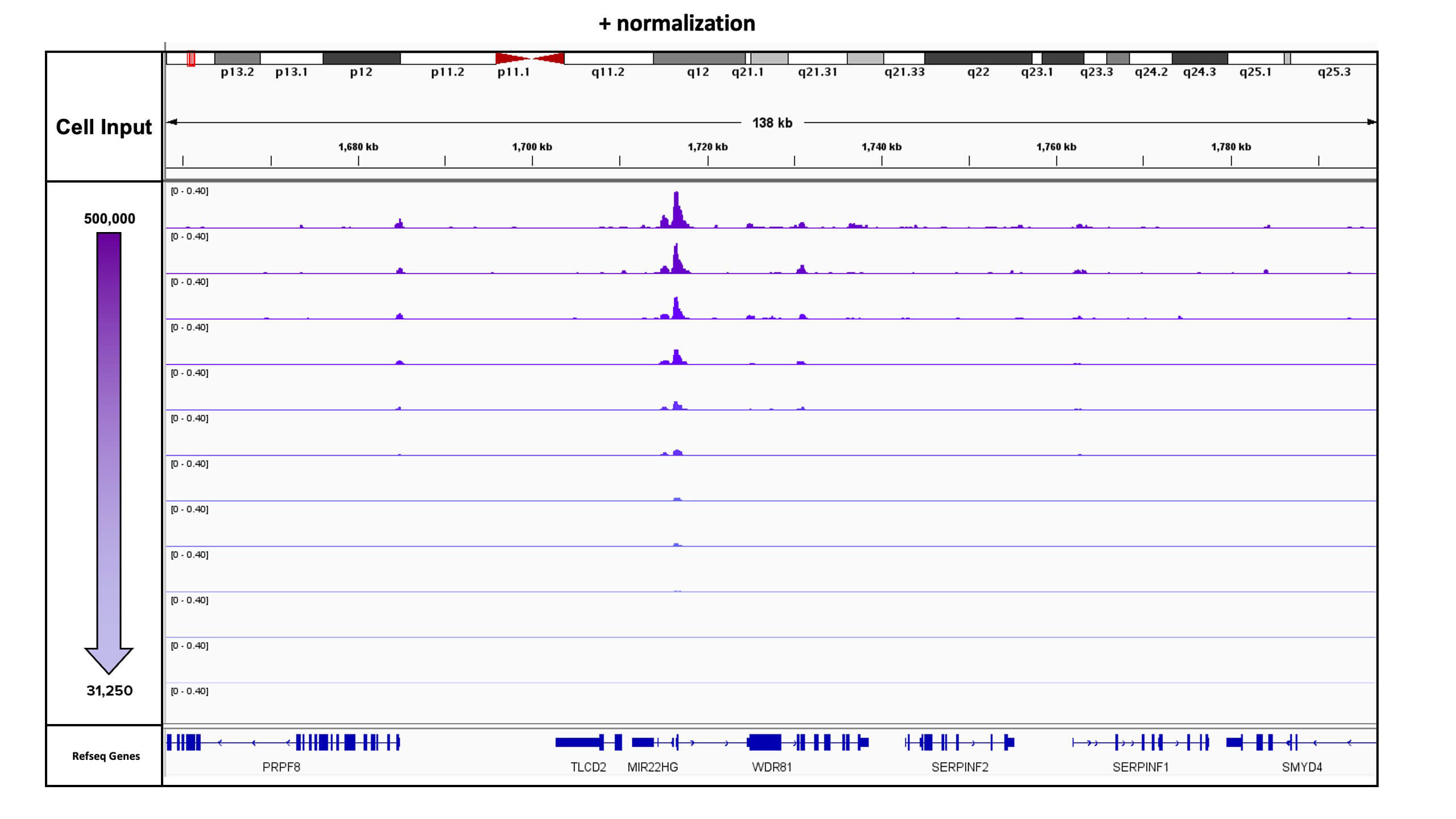
図2:異なる数のK562細胞に対し、CUT&Tag-IT R-loop Assayを実施した例(Spike-In標準化ありの場合)
この実験では、Spike-Inとして50,000個のショウジョウバエの細胞核を使用した。アッセイは500,000から31,250個の凍結保存K562細胞を用いてduplicationで実施した。この図は、duplicateで実施したすべてのサンプルの標準化ありのトラックを示す。ブラウザの示す遺伝子座はchr17:1,657,948-1,796,865である。
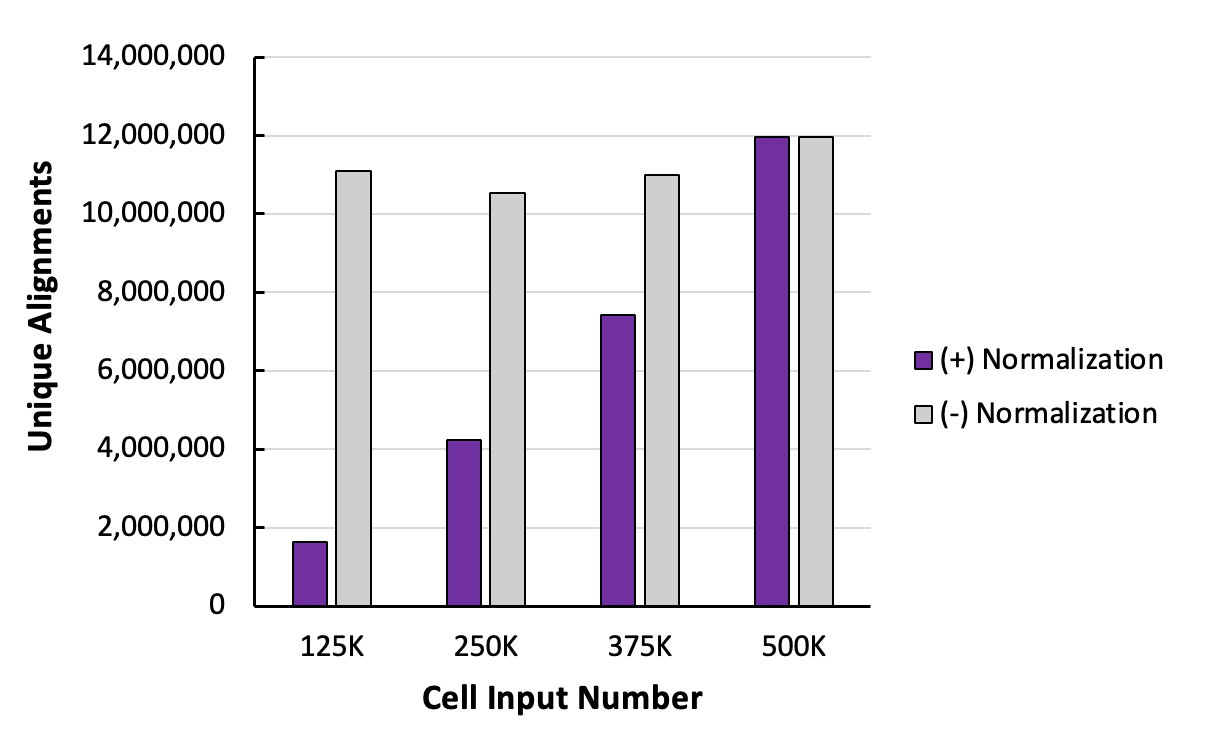
図3:K562細胞のユニークアラインメント
グラフは、4種の細胞数それぞれSpike-Inによる標準化なし(ー)と標準化あり(+)のヒトゲノムに対するユニークアラインメントである。各細胞数のグラフはduplicateで実施したうちの一方を示す。
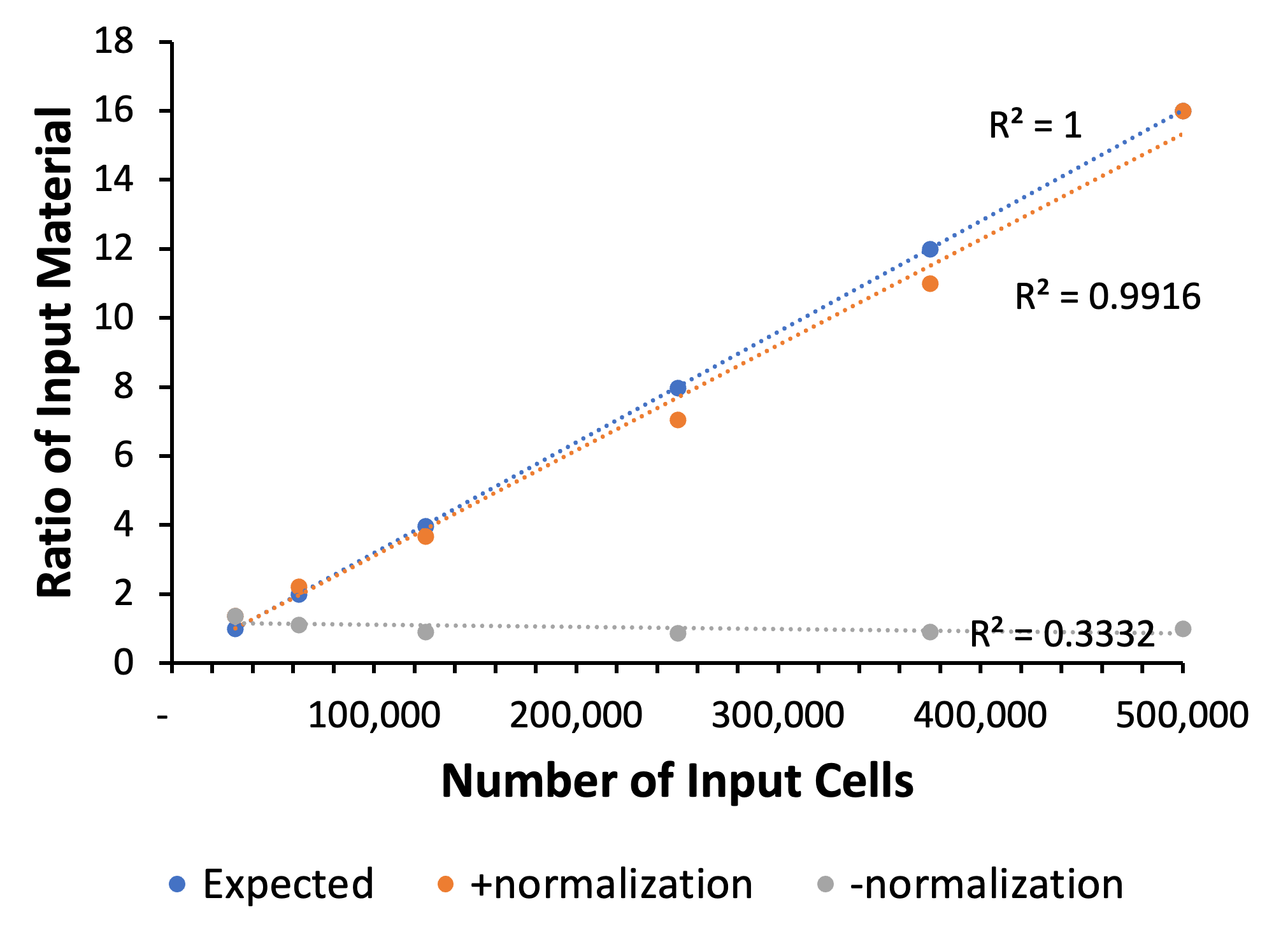
図4:標準化データと細胞数の相関関係
細胞数とリード数の比の関係を示す。標準化、および非標準化の各サンプルの値は、ヒトゲノムにマッピングした各サンプルのユニークフラグメント数を、drosophilaのユニークアライメント数で割って算出した。予測値(Expected)は、各サンプルの細胞数を最大数(50万)で割って算出した。
CUT&Tag-IT® Spike-In Control, R-loop の資料
こちらもご参照ください:
| Name | Format | Cat No. | Price | |
|---|---|---|---|---|
| CUT&Tag-IT® Spike-In Control, R-loop | 16 rxns | 53174 | ¥138,700 | Buy |