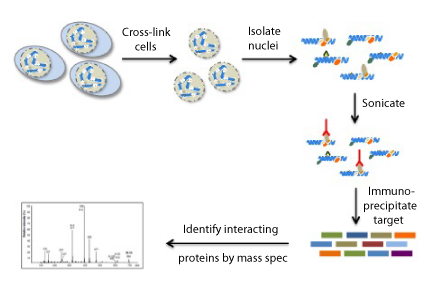
RIMEサービスの概要
RIME (Rapid Immunoprecipitation Mass spectrometry of Endogenous proteins) は、クロマチン免疫沈降と質量分析を組み合わせた、転写コファクターとクロマチン関連タンパク質を網羅的に同定するための強力な手法です。
RIMEサービスの内容:
- 核の単離およびソニケーション
- 免疫沈降
- タンパク質複合体の精製およびトリプシン消化
- 質量分析
- データ解析
RIMEでは何をするのか?
RIMEでは、ホルムアルデヒドによりクロスリンクされた細胞に対して免疫沈降を行い、質量分析法により標的タンパク質と相互作用するタンパク質を網羅的に同定します。
細胞をクロスリンクするメリット:
- 強力に架橋することにより、洗浄を強力に行えるため、非特異的な結合を低減
- 洗浄により分離してしまうような弱い結合タンパク質も同定可能
- 標的タンパク質とは直接相互作用しないが標的タンパク質と共に複合体を形成し、遺伝子発現を調節しているDNA結合タンパク質も捕捉可能
RIMEでわかること
- 転写因子、コファクター/共役因子の同定
- エピジェネティックな修飾関連タンパク質複合体を構成するタンパク質群の同定
- 相互作用が弱いタンパク質の同定
- タンパク質相互作用ネットワークのマッピング
- ChIP-Seqにより同定されたタンパク質を検証し、RIME標的タンパク質と相互作用する因子のゲノム上のマッピング
- 共役因子の遺伝子発現調節への役割について知見
- 隣接する部位における転写因子結合の顕著な共局在を同定。
What our customers are saying about us...
I have ordered RIME/ChIP-MS services. Since the target antibody was first tested by the Antibody Validation Service, I felt confident in proceeding with the main test. The technical staff explained the data and were willing to repeatedly answer my questions. This service provided me with valuable information and helpful support from all the staff members at Active Motif.
Naoko Hattori, PhD
Staff Scientist
Division of Epigenomics,
National Cancer Center Research Institute,
Tokyo, Japan
この度、RIME/ChIP-MSの受託サービスを利用しました。前試験で抗体性能をテストできたおかげで、自信をもって本試験を依頼できました。解析結果に関しては、詳細なご説明がいただけ、何回もの質問にも快く答えてもらえました。Active Motif社のスタッフの手厚いサポートのもと、信頼できる結果が得られる、とても有意義なサービスと感じました。
服部奈緒子 先生
国立がん研究センター研究所 エピゲノム解析分野
RIME受託サービスのデータ
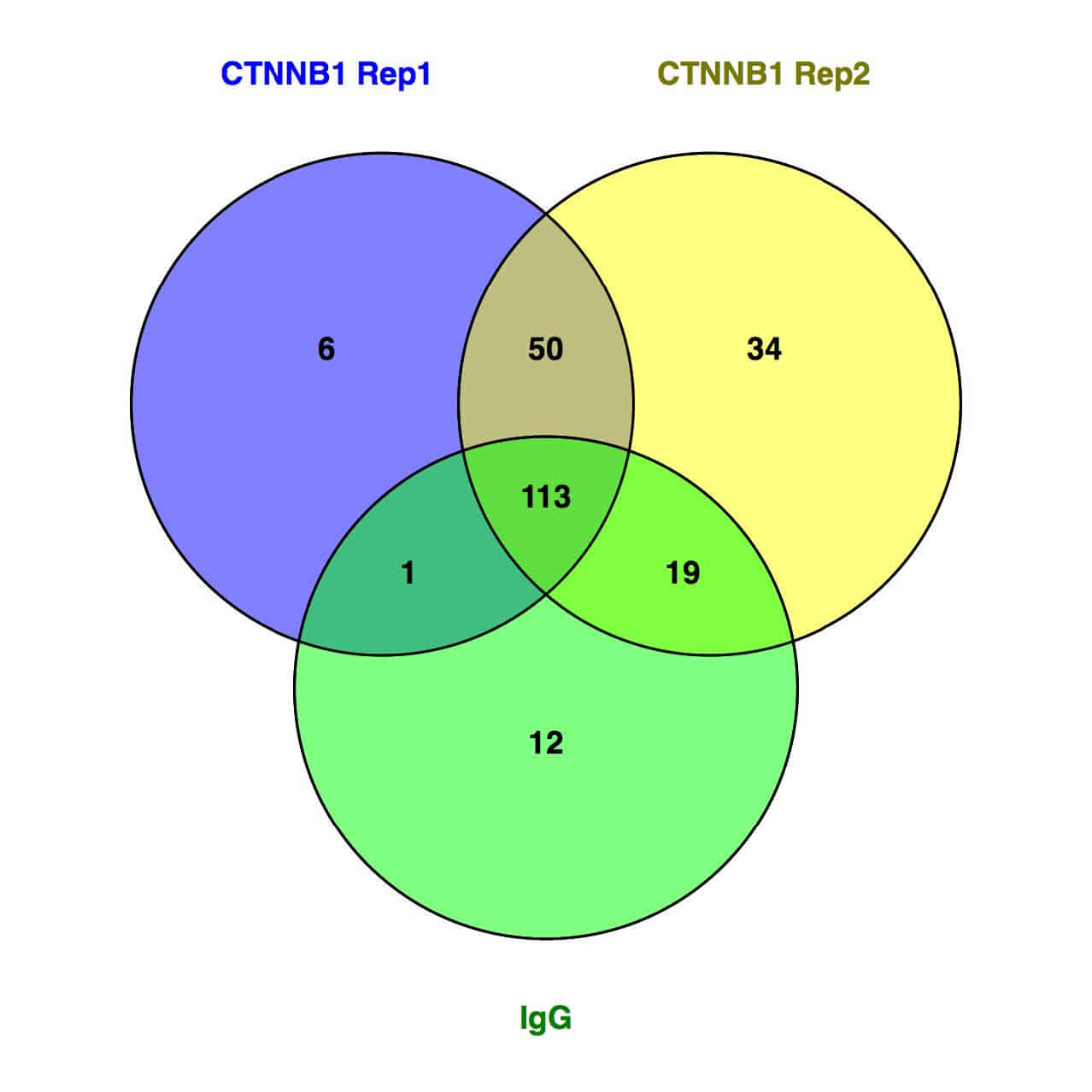
図1:β-カテニン相互作用タンパク質のベン図
SW480細胞において、ベイトタンパク質(β-カテニン)と相互作用するタンパク質を同定するためにRIMEを行った。β-カテニン(遺伝子名:CTNNB1)に対する抗体およびコントロールIgGを用いて免疫沈降を行い、回収したタンパク質を質量分析により解析した。ベン図は、β-カテニン抗体を用いた2サンプルとIgGで同定されたタンパク質のオーバーラップを示す。
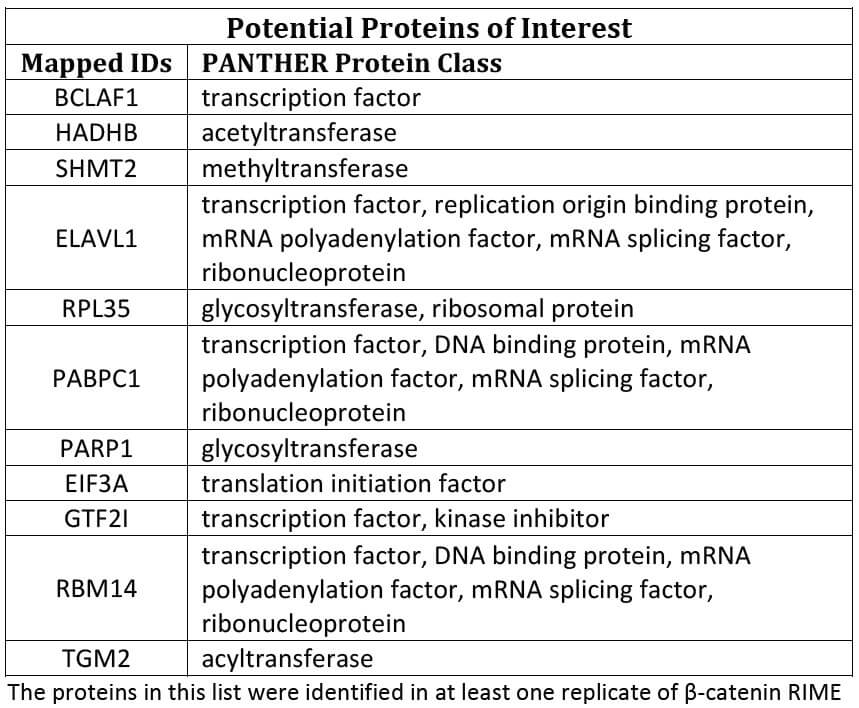
図2:標的タンパク質β-カテニンと相互作用するタンパク質の検出
この実験でβ-カテニンと相互作用することが見出されたタンパク質の上位11個をリストアップした。 ただし、ネガティブコントロールのIgGで検出されたタンパク質は除く。 このリストのタンパク質は、少なくとも1方のサンプルにおいて検出された。
図3: RIMEは濃縮されたタンパク質中の一般的な分子経路を特定できる。
β-カテニンと相互作用することが同定されたタンパク質のリストについて、PANTHERによる機能分類を行った。上位10個の遺伝子オントロジーの経路を同定し、p値に基づきランク付けした。
高度な応用例: RIMEとChIP-Seq
BRD4を標的タンパク質として、相互作用タンパク質のリストからRIMEを行いました。その結果、DNA topoisomerase 2Aが相互作用因子として同定されました。これは、リガンド依存性トポイソメラーゼがエンハンサーにリクルートされることを示した論文によって裏付けられています†。そこで、BRD4とTOP2Aそれぞれを標的としたChIP-Seqを行ったところ、共局在することが示されました。RIMEから得られたデータをChIP-Seqで拡張することにより、異なる転写因子を必要とする遺伝子サブセットを解明したり、RIMEで同定されたコファクターの同一遺伝子への共局在を確認したりすることができます。
図4 (左)および図5 (右):ChIP-Seqは、TOP2AとBRD4が互いの結合領域で共局在することを示した。
RIMEで同定されたBRD4相互作用タンパク質TOP2Aの結合部位をマッピングするためにChIP-Seqを行った。TOP2AとBRD4の結合部位が示されており、ゲノム上の多くの部位で共局在していることが確認された。図4は1番染色体上の領域、図5は3番染色体上の領域を示している。
参考文献
- †Puc, J. et al. Ligand-Dependent Enhancer Activation Regulated by Topoisomerase-I Activity, Cell. 2015 Oct 8;163(2):520-2.
RIME受託サービス使用文献
RIME受託サービスをご利用いただいたお客様の論文をデータベースから検索できます。
RIME受託サービスの資料
RIME Service Sample Submissionポータル
オンラインSample Submissionポータルは、解析したいサンプルの情報をアクティブ・モティフの担当者と共有し、依頼者には解析の進捗をご覧いただけるサイトです。 解析を依頼するために必要な連絡先、サンプルの情報、また解析の内容を記載していただきます。記載の方法や、サンプル提出方法について、ご不明な点がありましたら「[email protected]」までご連絡ください。
こちらもご参照ください:
| Name | Cat No. | 価格 (税抜) | |
|---|---|---|---|
| RIME | 25275 | Get Quote |
| Active Motif Epigenetic Services Brochure |
| RIME Services Sample Preparation |
| Epigenetic Services Citations |
質量分析の技術を利用したRIMEは、ホルムアルデヒドで固定化した細胞から、免疫沈降したタンパク質の結合タンパク質を網羅的に同定する手法です
細胞をクロスリンクするメリット:
- 強力な結合を形成することで、洗浄を強力に行えるため、非特異的な結合などのバックグラウンドの低減が可能
- 洗浄により分離してしまうような弱い結合タンパク質も同定可能
- 遺伝子調節に関与するが、標的タンパク質との直接相互作用によって機能しない隣接するDNA結合タンパク質も捕捉可能
RIMEでわかること:
- 転写因子、コファクター/共役因子の同定
- エピジェネティックな修飾関連タンパク質複合体の同定
- 結合が弱いタンパク質の同定
- タンパク質相互作用ネットワークのマッピング
- ChIP-Seqにより同定されたタンパク質を検証し、RIME標的タンパク質と相互作用する因子のゲノム上のマッピング
- 共役因子の遺伝子発現調節への役割について知見
- 隣接する領域における転写因子結合とともに起こる顕著な変化を同定
Figure 1: Venn Diagram of identified β-catenin interacting proteins for sample replicates and IgG
RIME was performed to identify proteins that interact with the bait protein (β-catenin) in SW480 [SW-480] cells. Immunoprecipitations were performed with an antibody against β-catenin (gene: CTNNB1). Immunoprecipitated proteins were measured by mass spectrometry. The Venn diagram shows the overlap of proteins identified for each replicate of β-catenin and anti-IgG RIME samples.
Figure 2: Detection of target protein β-catenin and potential proteins of interest
The top 11 proteins of interest that were found to interact with β-catenin in this experiment are listed, after removing proteins present in the IgG negative control. The proteins in this list were identified in at least one replicate of β-catenin RIME.
Figure 3: RIME identifies most prevalent molecular pathways among enriched proteins
PANTHER Functional Classification was performed for the list of proteins identified as interacting with β-catenin. The top ten gene ontology terms are identified and ranked by p-value.
応用例:RIME and ChIP-Seq
BRD4を標的タンパク質としてRIMEを実施し、相互作用のあるタンパク質としてTOP2Aが同定されました。そこで、実際にBRD4およびTOP2Aが、ゲノムDNA上で共局在することを確認するため、これらを標的とするChIP-Seqを実施しました。このように、ChIP-Seqを用いたRIMEデータの拡張は、異なる転写因子を必要とする遺伝子サブセットの解明または、RIMEで同定された補助因子が同一遺伝子のエンハンサー領域等に共局在することを明確にするのに役立ちます。


図1&図2:ChIP-SeqはTOP2AとBRD4の結合部位が共局在することを示す
RIMEにより同定された、BRD4と相互作用するタンパク質TOP2Aの結合サイトをマッピングするため、ChIP-Seqを行いました。TOP2AおよびBRD4の結合サイトを図に示します。両タンパク質はゲノム上の多くの部位で共局在することが確認されました。図の上段(図1)は1番染色体上の結合サイトを示し、下段(図2)は3番染色体の結合サイトを示します。