ChIC/CUT&RUN Assay Kitの概要
CUT&RUN検証済み抗体のご使用を推奨します。
CUT&RUN Spike-In Control: CUT&RUN反応で得られたサンプルデータ間における比較解析の信頼性を高める標準化試薬
DNA Library Prep Kit for Illumina®: ChIP-Seq、CUT&RUN、メチル化DNA (dsDNA)用のデュアルインデックスNGSライブラリー調製キット
Tissue Prep for NGS Assays: CUT&RUN用の組織サンプル前処理試薬
CUT&RUN (Cleavage Under Targets & Release using Nuclease) は、様々なクロマチン関連タンパク質とその修飾のゲノム全体にわたる分布を調べるために使用されるエピジェネティクスにおける解析手法です1-3。CUT&RUNはクロマチン免疫切断 (ChIC: Chromatin Immunocleavage) の派生手法です。CUT&RUNは、抗体を利用してクロマチン関連マークおよびタンパク質をターゲットにするという点でクロマチン免疫沈降 (ChIP) に似ていますが、ChIPよりも必要なサンプル量とシーケンス深度が少なくなっています。
CUT&RUNでは、無傷な細胞を用い、クロマチンに結合している標的タンパク質を、特異的な抗体でタグ付けします。次に、ミクロコッカスヌクレアーゼ (MNase) を使用して、目的タンパク質の結合部位で特異的にDNAを切断します。放出された断片を精製し、シーケンスして参照ゲノムにマッピングし、タンパク質の結合部位を決定します。ChIPとは異なり、CUT&RUNではアーティファクトの原因になり得るタンパク質とDNAの架橋が必要ありません。
CUT&RUNは、高感度で特異的であり、必要な細胞数がChIPよりも少ないため、クロマチン関連タンパク質を研究するための有用な手法です。そのため、転写因子やヒストン修飾などのクロマチン関連タンパク質の結合パターンをゲノムワイドに同定するのに理想的です。クロマチン関連タンパク質は、遺伝子発現、DNA複製、DNA修復、細胞分化などのさまざまな細胞プロセスの調節において重要な役割を果たします。これらのタンパク質の結合パターンを理解することで、これらの細胞プロセスがどのように制御されているかについての知見が得られます。
References:
1. Schmid, M. et al Mol Cell, 16(1): 147-157 (2004)
2. Skene, P.J. et al. (2017) Elife 6, e21856
3. Skene, P.J. et al. (2018) Nat. Protoc., 13, 1006-1019
ChIC/CUT&RUN Assay Kitの特長:
- 5,000 ~500,000個の細胞に適用可能
- 最適化されたプロトコルを備えた完全なキット
- ゲノムワイドなクロマチン関連タンパク質プロファイリングのために開発
ChIC/CUT&RUN Assay の手順

CUT&RUN vs. CUT&Tag vs. ChIP-Seq
| CUT&RUN | CUT&Tag | ChIP-Seq | |
|---|---|---|---|
| ネイティブ条件で実施? | はい | はい | いいえ |
| クロマチンの断片化法 | MNaseによる消化 | Tn5を用いたタグメンテーション | ソニケーション |
| 細胞数 | 500,000 cells | 5,000-500,000 cells | 1-10 million cells |
| シーケンス深度 * | 8 million reads † | 2 million reads ** | 20-50 million reads |
| ライブラリー調製キットの必要性 | あり。別工程で実施 | なし。タグメンテーションを利用 | あり。別工程で実施 |
| 適用可能な標的 | 幅広いヒストン修飾、転写因子およびコファクター | 主なヒストン修飾、いくつかの転写因子およびコファクター | 幅広いヒストン修飾、転写因子およびコファクター |
| 作業時間 | 1-2日 | 1-2日 | 2-3日 |
* Kaya-Okur et al. Nature Communications (2019) 10:1930
** For less abundant targets of interest, 8 to 10 million reads are recommended
† 25 million reads are recommended for transcription factor targets
ChIC/CUT&RUN Assay Kitの構成品
ChIC/CUT&RUN Assay Kitは、ドライアイス納品(-20℃保管の構成品)1箱と室温納品(4℃および室温保管の構成品)1箱の合計2箱で出荷されます。製品受領後、マニュアル記載の条件に従って保管してください。すべての試薬は適切に保管した場合、受け取り日から6か月間安定であることを保証します。
- Protease Inhibitor Cocktail, 500 µl, store at -20°C
- ChIC/CUT&RUN pAG-MNase, store at -20°C
- Glycogen 20 mg/ml, store at -20°C
- Histone H3K4me3 pAb, 10 µg, 1 mg/mL store at -20°C
- Negative Control IgG, Rabbit, store at -20°C
- 1 M Spermidine, store at -20°C
- Nuclei Isolation Buffer, store at 4°C
- Concanavalin A Beads, store at 4°C
- 0.1 M CaCl2, store at 4°C
- Stop Solution, store at 4°C
- 1X Binding Buffer, store at 4°C
- Dig-Wash Buffer, store at 4°C
- 0.5 M EDTA, store at Room temperature
- DNA Purification Wash Buffer, store at Room temperature
- DNA Purification Elution Buffer, store at Room temperature
- DNA Purification Binding Buffer, store at Room temperature
- 3M Sodium Acetate, store at Room temperature
- DNA Purification Columns SF, store at Room temperature
- 0.2 mL Strip Tubes, Attached Caps, store at Room temperature
- RNase A, store at -20°C
- 5% Digitonin, store at -20°C
ChIC/CUT&RUN Assay Kitのデータ
図1. K562細胞におけるヒストンマークのCUT&RUNプロファイリング。
アクティブ・モティフのH3K27ac抗体 (Cat. No. 91193)、H3K4me3抗体 (Cat. No. 91263)、H3K27me3抗体 (Cat. No. 39155) を用いた、K562細胞50万個におけるCUT&RUNのピークトラックをIGBブラウザーで示した。
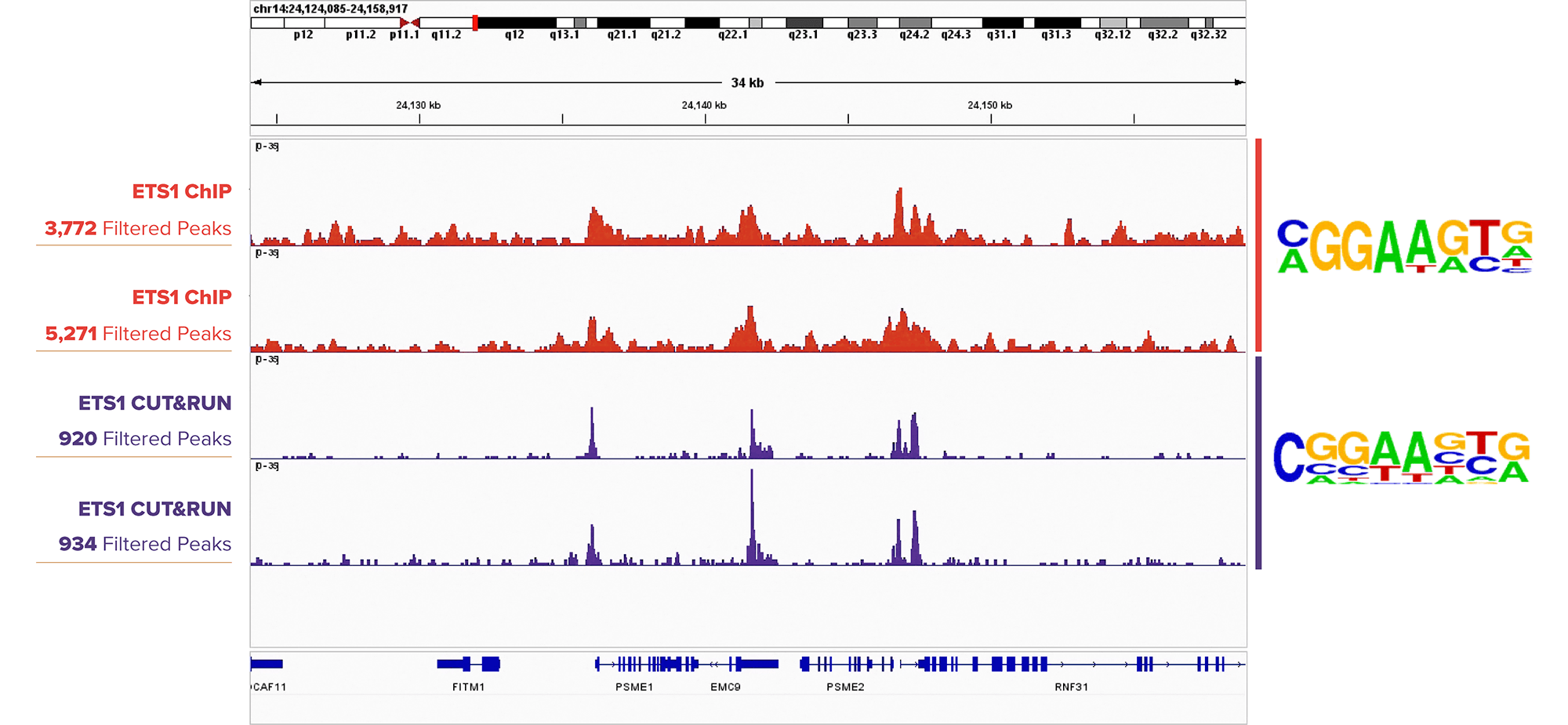
図2. ETS1 CUT&RUNピークプロファイルはETS1 ChIP-Seqプロファイルと類似
ETS1抗体 (アクティブ・モティフ Cat. No. 39580) を用いたDIPTG細胞に対するChIP-Seqアッセイ (上の2トラック) とK562細胞に対するCUT&RUNアッセイ (下の2トラック) のIGBブラウザーのピークトラック。Homer解析により得られた配列のロゴ (トラックの右側) は、両方のアッセイ群においてETS1モチーフが濃縮されていることを示している。
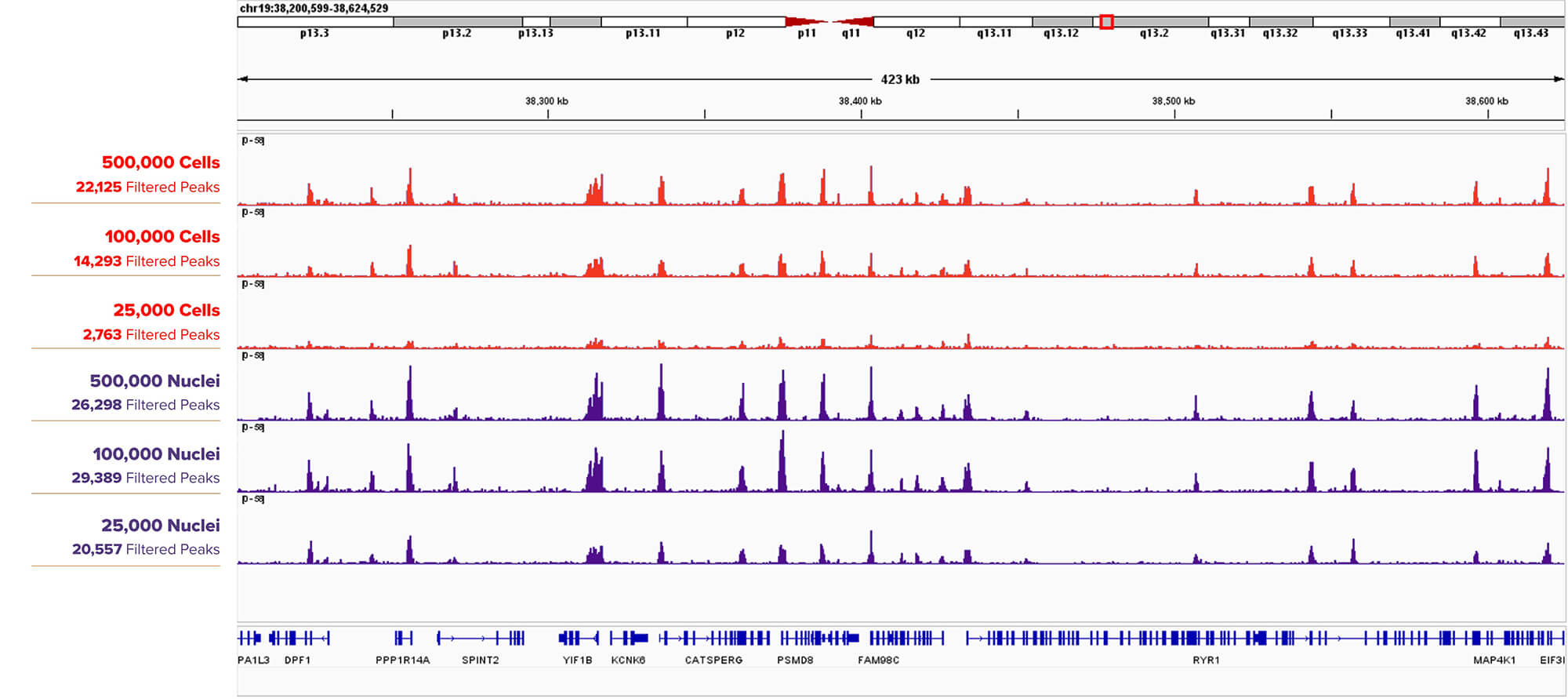
図3. H3K4me3 抗体を用いたCUT&RUNの結果(細胞または核から行ったときの比較)
500,000個、100,000個、25,000個のK562細胞(上3つのトラック)またはその核(下3つのトラック)に対し、アクティブ・モティフのH3K4me3抗体 (Cat. No. 39060) を用いてCUT&RUNアッセイを実施した。Filtered Peakは、細胞よりも核を用いた場合の方が少ないサンプル量でも高い値を示し、サンプル量が多い場合においても、核を用いた方がより高いピークを示している。
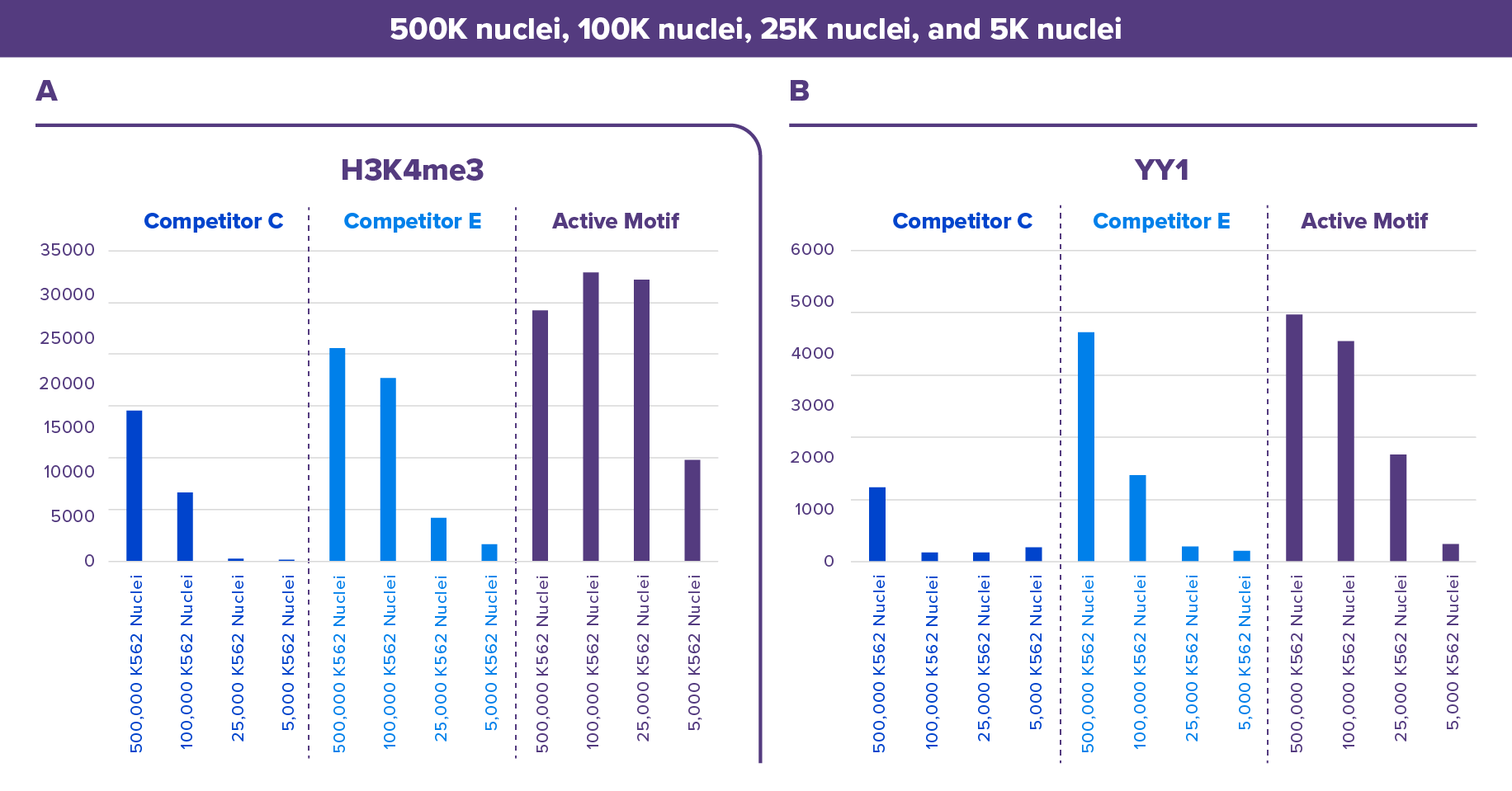
図4. アクティブ・モティフのCUT&RUNキットは他社競合製品より高感度
H3K4me3 (アクティブ・モティフCat. No. 39916) およびYY1 (アクティブ・モティフCat. No. 61980) について、アクティブ・モティフ、競合C社、競合E社のキットを用いてCUT&RUNを実施した。K562細胞の核500,000個、100,000個、25,000個、5,000個で得られたピークスコアを棒グラフで示す。
図5. アクティブ・モティフのCUT&RUNキットは、5,000個の核からのヒストンマークの検出に成功
H3K4me3抗体 (アクティブ・モティフ Cat. No. 91263) と、アクティブ・モティフ、競合E社、競合C社のキットを用いて、K562細胞の核5,000個に対してCUT&RUNを実施した。アクティブ・モティフのCUT&RUNキットは、競合品よりもロバストな結果が得られた。
図6. アクティブ・モティフのCUT&RUNキットは、25,000個の核からの転写因子結合サイトの検出に成功
YY1抗体 (アクティブ・モティフ Cat. No. 61980) と、アクティブ・モティフ、競合E社、競合C社のキットを用いて、K562細胞の核25,000個に対してCUT&RUNを実施した。アクティブ・モティフのCUT&RUNキットは、競合品よりもロバストな結果が得られた。
ChIC/CUT&RUN Assay KitのFAQ
核ペレットが見えませんが採れているでしょうか?
ペレットは見えないかもしれません。遠心後にペレットがチューブのどの位置にあるかわかるように、遠心機にチューブを入れるときの向き(キャップの位置など)を決め、次のステップを注意深く進めてください。
Concanavalin A (Con A) ビーズの使い方について教えてください。また、細胞へ結合させた場合にどのように見えますか?
この動画では、CUT&RUNまたはCUT&Tagアッセイを行う際のCon Aビーズの調製方法と、ビーズ単体または細胞や核と結合させた場合に、Con Aビーズがどのように観察されるかなどを紹介しています。さらに、全体を通じたチェック項目もご覧いただけます。
推奨されるシーケンス深度を教えてください。
転写因子の場合は2,500万リードを推奨しますが、ヒストンマークの場合は、200万リードまで下げることができます。
CUT&RUN Assay Kitを使用するときのおすすめのリード長は?
38塩基長のペアエンド (PE38) を推奨しています。
転写因子を標的とする場合は何個の細胞を使うべきですか?
細胞は50万個を使用し、CUT&RUN検証済みの抗体を用いることを推奨します。
データに高いバックグラウンドがあるのはなぜですか?
必ず生細胞を用いて開始してください。Countess IIのような自動セルカウンター、またはトリパンブルー染色と血球計算盤を用いて、細胞/核が無傷であることを確認してください。
ネガティブコントロールのライブラリーは作れますか?
はい。Negative Control IgGを使用してライブラリーが作成できます。
SPRIビーズがペレット化しない、またはペレット化に時間がかかりすぎます。どうしたら良いでしょうか?
まず、磁石を使ってSPRIビーズをチューブ上方に集め、徐々にチューブの下方へと下げていきます。その後、チューブをマグネットプレートの上に置いてください。
CUT&RUNについて論文実績のあるアクティブ・モティフの抗体はありますか?
こちらから論文実績のあるアクティブ・モティフの抗体をご確認いただけます。ただし、アクティブ・モティフでは検証を行っていません。
ポジティブコントロール抗体を使用した場合のライブラリートレースの例はありますか?
以下にポジティブコントロールのライブラリートレースを4例示します。




ネガティブコントロール抗体を使用した場合のライブラリートレースの例はありますか?
以下にネガティブコントロールのライブラリートレースを2例示します。


CUT&RUNの結果は、細胞サンプルと核サンプルではどのような違いがありますか?
核の使用を推奨しますが、転写因子の標的領域によっては細胞サンプルの方がピークプロファイルが高い場合もあります。詳細は以下の図をご参照ください。
K562細胞について、500,000個の細胞と核を、SUZ12抗体(アクティブ・モティフ Cat No. 39357)を用いてCUT&RUNをそれぞれ実施した。細胞サンプルと核サンプルからは非常によく似た結果が得られた。比較のため、HEPG2細胞からのChIP-seqデータを上(緑)で示す。
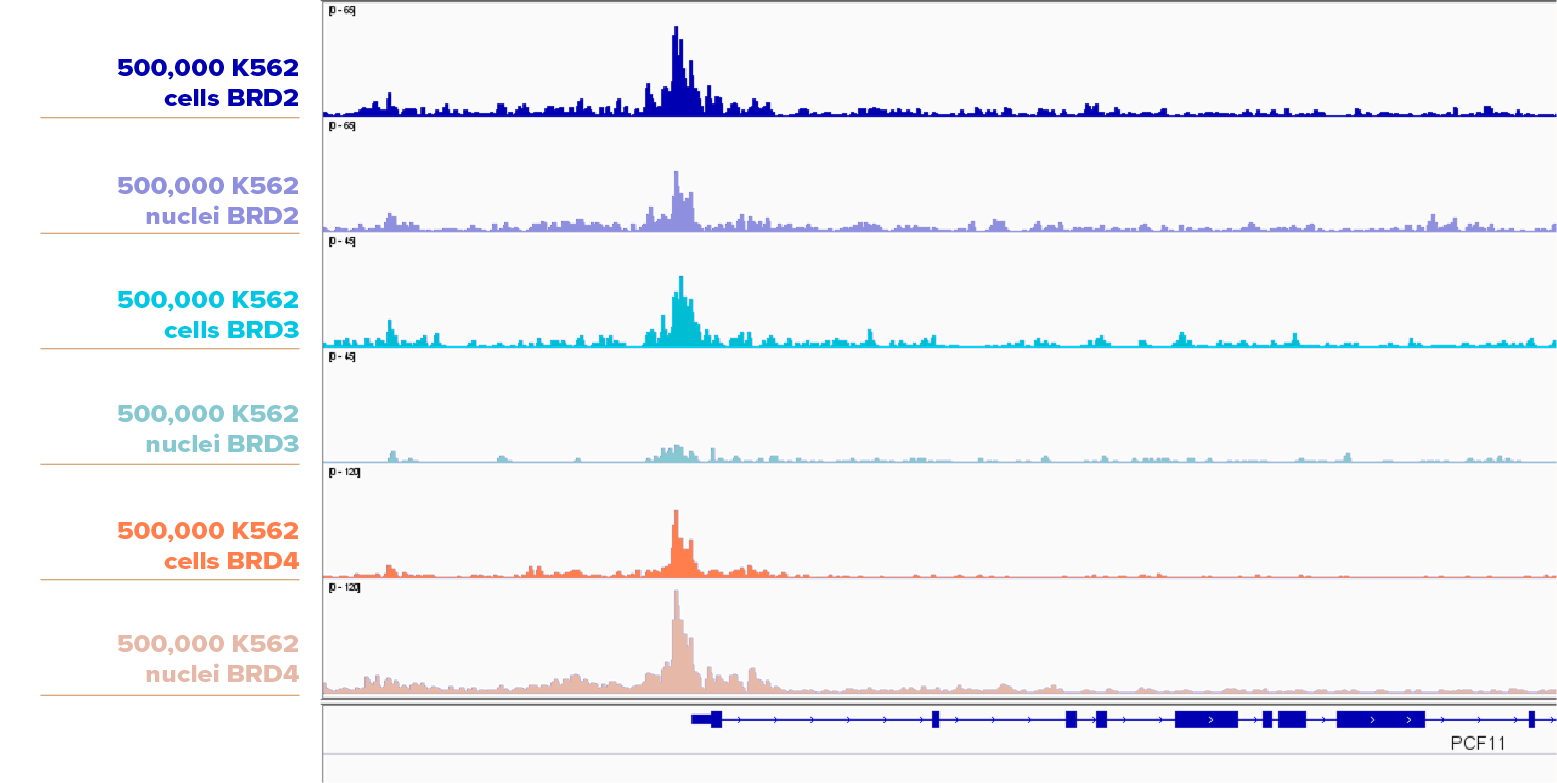
K562細胞について、500,000個の細胞と核を、BRD2抗体 (アクティブ・モティフ 61797)、BRD3抗体 (アクティブ・モティフ Cat No. 61489)、 BRD4 (アクティブ・モティフ Cat. No. 91301) を用いてCUT&RUNをそれぞれ実施した。 BRD2およびBRD3については、細胞サンプルの方が核サンプルよりもわずかに高いピークを示したが、BRD4については、核サンプルの方が細胞サンプルよりも高いピークを示した。
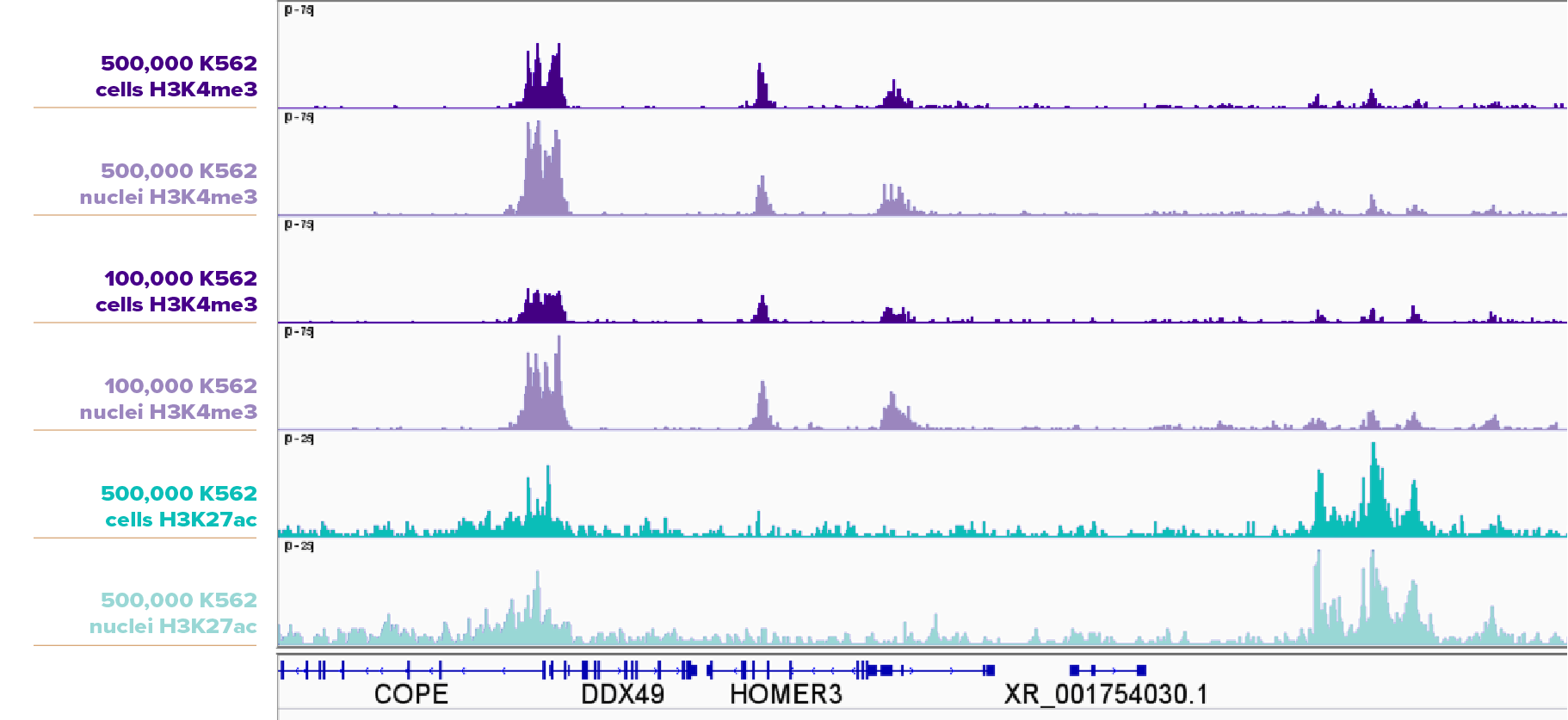
K562細胞について、500,000個、100,000個の細胞と核を、H3K4me3抗体 (アクティブ・モティフ Cat No. 91263) を用いてCUT&RUNを実施し、 500,000個の細胞と核を、H3K27ac抗体 (アクティブ・モティフ Cat No. 91193) を用いてCUT&RUNを実施した。それぞれ、核サンプルの方が細胞サンプルよりも高いピークを示した。
ChIP-SeqやCUT&Tagで検証された抗体は、CUT&RUNで機能しますか?
CUT&RUN、CUT&Tag、ChIP-Seqはワークフローが全く異なります。ある抗体がCUT&TagやChIP-Seqで機能しても、CUT&RUNで機能するとは限りません。CUT&RUNには、弊社のCUT&RUN検証済み抗体を使用することを推奨します。
YY1抗体 (pAb) を使用した場合のライブラリートレース例はありますか?
JunD抗体 (pAb) を使用した場合のライブラリートレース例はありますか?
ChIC/CUT&RUN Assay Kitの資料
ChIC/CUT&RUN Assay Kit Manual [Japanese]
ChIC/CUT&RUN Assay Kit Manual [English]
Counting Cells and Nuclei for Epigenetic Applications
こちらもご参照ください:
ChIC/CUT&RUN provided under license from EpiCypher, under US Patent No. 7790379, 11885814 and related patents and pending applications.