CUT&RUN Spike-In Controlの概要
CUT&RUN (Cleavage Under Targets & Release Using Nuclease)は、様々なクロマチン関連タンパク質とその修飾のゲノムワイドな分布を調べるための強力な手法として登場しました。しかし、グローバルな修飾変化が起こった場合、データセット間の差異を同定することは困難です。さらに、サンプル量の測定が不正確だったり、作業中の手技にばらつきが生じたりすると、サンプルデータ間のばらつきが大きくなります。このような場合、現在利用可能なバイオインフォマティクスに基づく標準化手法は適用でません。サンプル量や手技の偏り、ばらつきを克服するための唯一の信頼できる方法は、すべてのサンプルに既知の標準物質を添加(“spike-in”)することです。アクティブ・モティフはこれまで、ChIP-SeqとCUT&Tag用のspike-in標準化のための試薬を提供してきましたが、今回CUT&RUNにも同様のアプローチを導入しました。
アクティブ・モティフのCUT&RUNにおける標準化法では、CUT&RUNの前に、凍結したショウジョウバエ由来の細胞核をサンプルに添加(“spike-in”)します。その後、抗体反応において標的に対する抗体とショウジョウバエのH2Avに対する抗体を混合してインキュベーションします。この抗H2Av抗体が、全サンプルで同様にショウジョウバエヒストンに特異的に結合し、ショウジョウバエゲノムを断片化に導きます。シーケンス後、ショウジョウバエのピークに基づいて標準化係数を算出し、標的サンプルのゲノムのトラックに適用します。このCUT&RUN Spike-In法により、使用する標的用抗体に依存せず、バイアスのないCUT&RUNデータの標準化が可能になります。
このCUT&RUN Spike-In Controlは、ChIC/CUT&RUN Assay Kit (Catalog No. 53180)にご使用いただけます。
CUT&RUN Spike-In Controlの特長
- CUT&RUNデータセット間の違いを同定
- 細胞数の違いなどにより隠されたサンプル間の違いも解明
- CUT&RUNを実施する際、サンプルにSpike-In Nucleiを加え、標的の抗体とSpike-In抗体を同時に使用するだけ
CUT&RUN Spike-In Controlの構成品
- CUT&RUN Spike-In Antibody, store at -20°C
- CUT&RUN Spike-In Nuclei, store at -80°C
CUT&RUN Spike-In Controlのデータ
アクティブ・モティフの標準化法は1、哺乳動物サンプルとの交差性がないSpike-In Antibodyを用いているため、おそらくほとんどすべての哺乳動物に対するCUT&RUN反応に適用可能です。CUT&RUN反応に使用する凍結保存されたDrosophilaの核とSpike-In Antibodyの量は、 Drosophilaのリードが全シーケンスリードの5-10%程度となることを目標とした最適化が必要かもしれません。ただし、H3K4me3のような局在性の強いヒストン修飾に対する抗体を使用する場合は、Spike-In Nuclei : テストサンプル細胞の比率を1 : 20にすることを推奨します。H3K27me3のような拡がりをもつマークに対しては、Spike-In Nuclei : テストサンプル細胞の比率を1:10にすることを推奨します。YY1などの転写因子に対しては、Spike-In Nuclei : テストサンプル細胞の比率は1 : 100を推奨します。
このアプローチの有用性を示すため、使用する細胞数を変えてCUT&RUN反応を実施し、ヒストン修飾レベルの違いを調べました。様々な数の凍結保存ヒトK562細胞(500,000、400,000、300,000、200,000、100,000、50,000)を、凍結保存Drosophilaの核20,000個(H3K4me3の場合) (図1)または10,000個(YY1の場合) (図2)と組み合わせて各実験を行いました。
各実験は重複アッセイとし、H3K4me3およびYY1に対し、CUT&RUN Spike-Inの有効性を評価しました。ライブラリーを定量し、サンプルあたり2,000万~3,000万リードの深さでシーケンスを実施しました。しかし、各サンプルのリード数が同じになるようにシーケンスすると、サンプル量の違いはマスクされてしまいます(図3および4)。したがって、サンプル量の違いを表すためにはspike-in標準化を行うことが必要となります2。標準化には、Drosophilaのリード数が最も少ないサンプルを用いてサンプル間の標準化係数を求め、それを各サンプルのヒトのリード数に適用しました。標準化されたヒトのリード数を得た後、一般的なCUT&RUN解析パイプラインを使用してbigWigのピークコールを行いました。標準化を適用した結果、サンプル量の比率に対して予想される結果と相関がみられました(図5および6)。
References
- Egan, B. et al. (2010) PLoS ONE. 11(11): e0166438
- Taruttis et al. (2017) Biotechniques 62:53-61
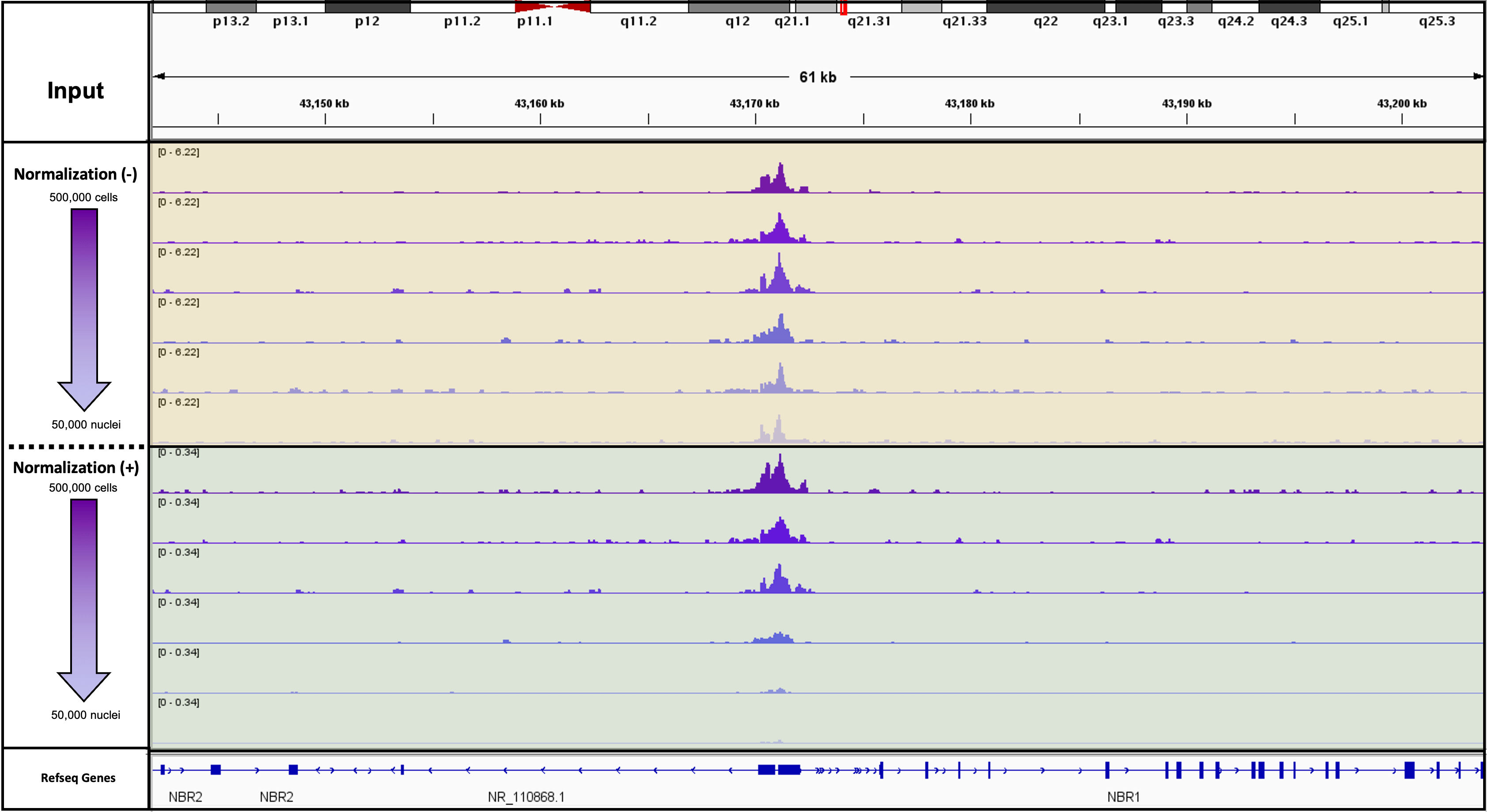
図1: H3K4me3を標的としたCUT&RUNを実施したときの細胞数とSpike-in標準化の有無の関係
20,000個のSpike-In Nucleiを異なる数のK562細胞(500,000個、400,000個、300,000個、200,000個、100,000個、および50,000個)に添加して、CUT&RUNアッセイを実施した。標準化の有無による結果を示す。
図2: YY1を標的としたCUT&RUNを実施したときの細胞数とSpike-in標準化の有無の関係
5,000個のSpike-In Nucleiを500,000個、400,000個、300,000個、200,000個、100,000個、および50,000個のK562細胞に添加し、CUT&RUNアッセイを実施した。標準化の有無による結果を示す。
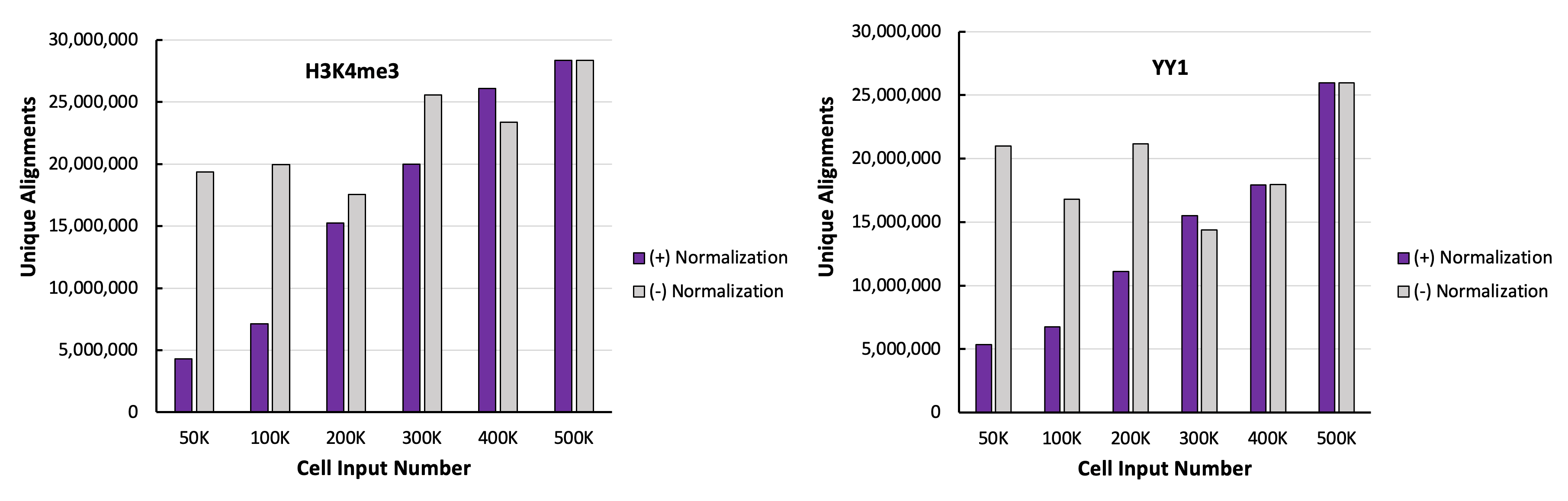
図3: 標準化の有無とK562細胞のペアアラインメントの関係
標準化なしの場合、マッピングされたリード数は、H3K4me3 (左)とYY1 (右)を標的としたいずれの実験においても細胞数の違いが反映されていない。このことから、標準化が必要であることがわかる。
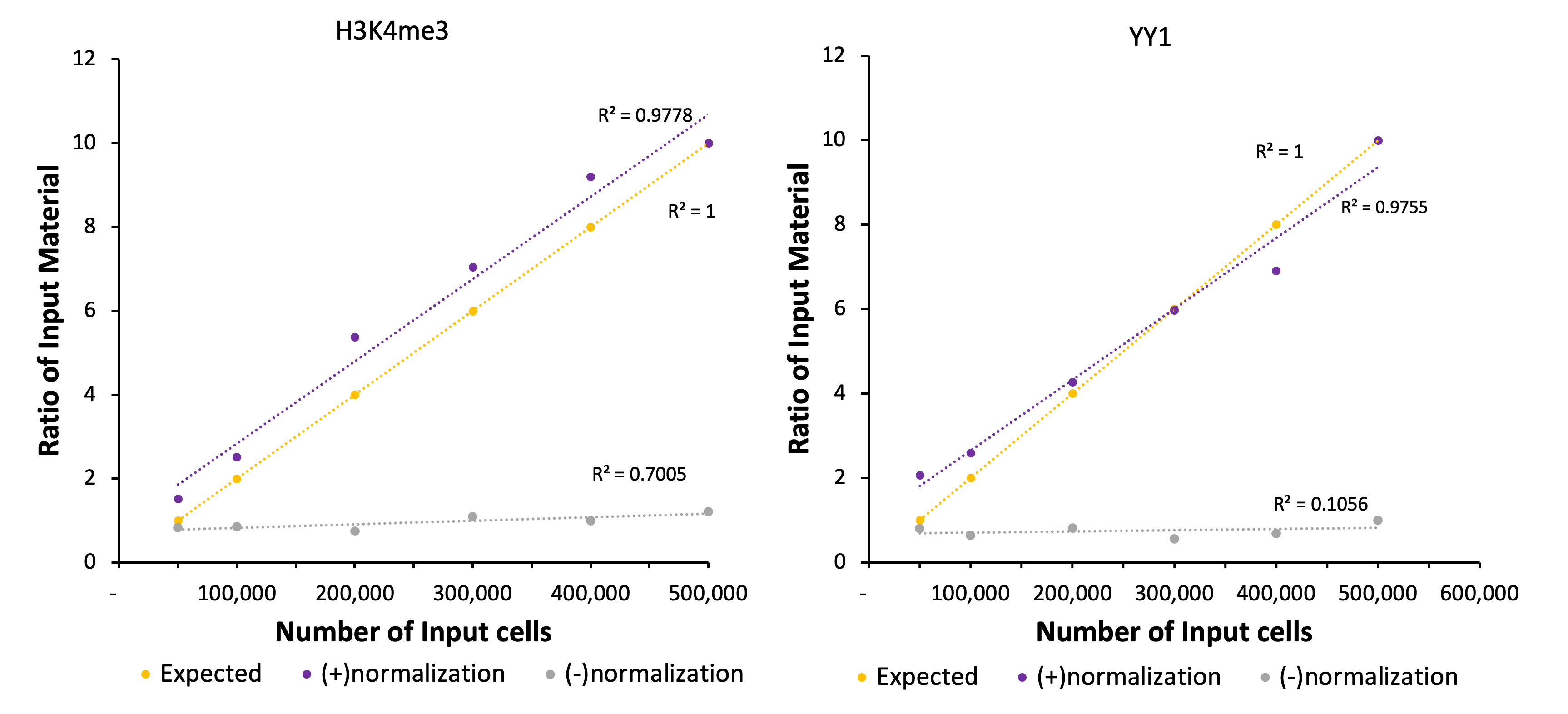
図4: Drosophilaのペアアラインメントによる標準化の有無とK562細胞のペアアラインメントの関係
各細胞数のK562細胞のペアアラインメントを、50万個のアッセイで得られたDrosophilaのアラインメント数で標準化した。これにより、H3K4me3 (左)とYY1 (右)のいずれも、細胞数に基づいた期待されるリード数の比率が回復した。
CUT&RUN Spike-In Controlの資料
こちらもご参照ください:
| Name | Format | Cat No. | 価格 (税抜) | |
|---|---|---|---|---|
| CUT&RUN Spike-In Control | 24 rxns | 53183 | $US 695 | Add to Cart |